Desambiguación de agentes del sector I+D+i
Desarrolladores
El proyecto ha sido desarrollado por el grupo Elhuyar y coordinado por Xabier Saralegi.
Resumen
El proyecto tiene como objetivo el desarrollo e implementación de componentes software para reconocer y desambiguar los investigadores y organismos de las bases de datos de ayudas públicas a la ciencia y la innovación, y de sus resultados asociados: publicaciones científicas y patentes
La necesidad del proyecto se justifica por el hecho de que un mismo agente del sector I+D+i puede aparecer generalmente con diferentes denominaciones en las bases de datos empleadas en el proyecto de Inteligencia Competitiva, por lo que para su caracterización resulta necesario localizar todas las entradas relativas a dicho agente en todas las bases de datos disponibles (e.g.: proyectos de investigación en los que ha participado, artículos que ha coautorizado, etc).
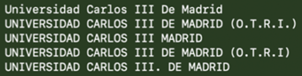
La desambiguación tiene lugar, en primer lugar, para cada base de datos, ya que incluso dentro de una misma base de datos un agente (investigador u organismo) puede aparecer con diferentes nombres e identificadores. En segundo lugar, el desarrollo SW busca todas las entradas de un mismo agente en diversas bases de datos.
En resumen: se trata de identificar de manera única a cada investigador u organismo del sector I+D+i español, y localizar las entradas relativas a dicho agente en todas las bases de datos. Esta desambiguación resulta imprescindible como paso previo a la caracterización de los agentes del sector.
El sistema desarrollado consta de dos módulos:
- Módulo de agrupación: Localiza pares de nombres que son podrían corresponder a la misma entidad. La búsqueda de pares de candidatos a pertenecer a una misma entidad se realiza únicamente en base al nombre aplicando las siguientes técnicas:
- Normalización de nombres: se armonizan abreviaturas, iniciales, tildes, orden los elementos del nombre, etc.
- String-matching: Cálculo eficiente de distancias entre todos los pares de nombres, identificando como candidatos aquellos cuya distancia sea inferior a un umbral
- Modulo de desambiguación: Explota los metadatos y el texto no estructurado disponible para decidir si cada par identificado por el módulo anterior debe o no fusionarse.
- Clasificación supervisada: Entrenamiento de un clasificador basado en redes neuronales y Gradient Boosting cuya salida establece si los agentes de cada par de candidatos son o no el mismo.
- Construcción de clases de equivalencia: Mediante clustering aglomerativo se agrupan todas las instancias que corresponden a cada agente único. A continuación se muestra el diagrama del desarrollo para la desambiguación de agentes del Sector de I+D+i:
Input: Dumps de Parstat, PN, Cordis y Scopus
Agrupación de pares candidatos de equivalentes de organizaciones y autores Extracción de nombres de organizaciones y autores Normalización de nombres String-maching entre nombres normalizados de mismos (intra) y distintos cospus (inter) Output/Input: Pares candidatos de organizaciones y autores equivalentes
Desambigüación de pares candidatos y contrucción de clases de equivalencia Desambigüación de pares candidatos mediante clasificación supervisada
Construcción de clases de equivalencia mediante clustering supervisado Asignación de id canónico
Output: Organizaciones y autores equivalentes
La evaluación del sistema ha permitido concluir la importancia de esta tarea como paso previo a la caracterización de agentes, ya que la búsqueda de pares de candidatos proporciona un muy elevado número de potenciales duplicados (en algunos casos superior incluso al 50% de las entradas de la base de datos). Las prestaciones de los clasificadores desarrollados se han medido mediante el etiquetado manual de conjuntos de datos, permitiendo establecer una precisión de alrededor del 90% para el caso de los investigadores y del 75% para organizaciones.
A continuación se muestran los pares de entradas de candidatos identificados por el módulo de agrupamiento en las diversas bases de datos:
| Autores | ||
|---|---|---|
| Parejas enlazadas | % Autores enlazados | |
|
Intra-corpus |
||
| Patstat-Patstat | 232.782 | 65% |
| PN-PN | 28.760 | 13.2% |
| Scopus-Scopus | 300.658 | 52.5% |
|
Inter-corpus |
||
| PN-Patstat | 96.719 | 16.9% |
| PN-Scopus | 264.413 | 33.6% |
| Patstat-Scopus | 181.248 | 19.5% |
| Organizaciones | ||
|---|---|---|
| Parejas enlazadas | % Organizaciones enlazados | |
|
Intra-corpus |
||
| Patstat-Patstat | 54.407 | 79,7% |
| PN-PN | 2.407 | 18,7% |
| Scopus-Scopus | 61.765 | 79,2% |
| Cordis-Cordis | 4.184 | 31,2% |
|
Inter-corpus |
||
| PN-Patstat | 27.322 | 33,7% |
| PN-Scopus | 16.351 | 18,0% |
| PN-Cordis | 6.187 | 27,2% |
| Patstat-Scopus | 33.433 | 22,9% |
| Patstat-Cordis | 19.061 | 24,4% |
| Scopus-Cordis | 15.380 | 17,5% |

